本教程描述了如何读取和绘制传感器位置,以及MNE-Python如何处理传感器的物理位置。像往常一样,我们将从导入我们需要的模块开始:
1 | from pathlib import Path |
关于montage和layout(蒙太奇和传感器布局)
montage包含3D传感器位置(x, y, z以米为单位),可以分配给现有的EEG/MEG数据。通过指定传感器相对于大脑的位置,蒙太奇在计算正向解和逆估计中起着重要作用。
相反,layout是传感器位置的理想化二维表示。它们主要用于在拓扑图中排列单个传感器子图,或用于显示上面观的传感器的近似相对排列。
注意,如果专门处理eeg数据,使用montage更好。理想化的montage,例如由制造商提供的那些,通常被称为template montage(模板)。
使用内置的montage
MEG传感器的三维坐标包含在MEG系统的原始记录中。它们在加载时自动存储在Raw对象的info属性中。而由于头部形状的不同,eeg电极的位置变化很大。许多EEG系统的模板montage都包含在MNE-Python中,可以通过使用mne.channels. get_builtin_montage()来获取:
1 | builtin_montages = mne.channels.get_builtin_montages(descriptions=True) |
standard_1005: Electrodes are named and positioned according to the international 10-05 system (343+3 locations)
standard_1020: Electrodes are named and positioned according to the international 10-20 system (94+3 locations)
standard_alphabetic: Electrodes are named with LETTER-NUMBER combinations (A1, B2, F4, …) (65+3 locations)
standard_postfixed: Electrodes are named according to the international 10-20 system using postfixes for intermediate positions (100+3 locations)
standard_prefixed: Electrodes are named according to the international 10-20 system using prefixes for intermediate positions (74+3 locations)
standard_primed: Electrodes are named according to the international 10-20 system using prime marks (‘ and ‘’) for intermediate positions (100+3 locations)
biosemi16: BioSemi cap with 16 electrodes (16+3 locations)
biosemi32: BioSemi cap with 32 electrodes (32+3 locations)
biosemi64: BioSemi cap with 64 electrodes (64+3 locations)
biosemi128: BioSemi cap with 128 electrodes (128+3 locations)
biosemi160: BioSemi cap with 160 electrodes (160+3 locations)
biosemi256: BioSemi cap with 256 electrodes (256+3 locations)
easycap-M1: EasyCap with 10-05 electrode names (74 locations)
easycap-M10: EasyCap with numbered electrodes (61 locations)
easycap-M43: EasyCap with numbered electrodes (64 locations)
EGI_256: Geodesic Sensor Net (256 locations)
GSN-HydroCel-32: HydroCel Geodesic Sensor Net and Cz (33+3 locations)
GSN-HydroCel-64_1.0: HydroCel Geodesic Sensor Net (64+3 locations)
GSN-HydroCel-65_1.0: HydroCel Geodesic Sensor Net and Cz (65+3 locations)
GSN-HydroCel-128: HydroCel Geodesic Sensor Net (128+3 locations)
GSN-HydroCel-129: HydroCel Geodesic Sensor Net and Cz (129+3 locations)
GSN-HydroCel-256: HydroCel Geodesic Sensor Net (256+3 locations)
GSN-HydroCel-257: HydroCel Geodesic Sensor Net and Cz (257+3 locations)
mgh60: The (older) 60-channel cap used at MGH (60+3 locations)
mgh70: The (newer) 70-channel BrainVision cap used at MGH (70+3 locations)
artinis-octamon: Artinis OctaMon fNIRS (8 sources, 2 detectors)
artinis-brite23: Artinis Brite23 fNIRS (11 sources, 7 detectors)
brainproducts-RNP-BA-128: Brain Products with 10-10 electrode names (128 channels)
这些内置的EEG montage可以通过mne.channels. make_standard_montage加载:
1 | easycap_montage = mne.channels.make_standard_montage("easycap-M1") |
<DigMontage | 0 extras (headshape), 0 HPIs, 3 fiducials, 74 channels>
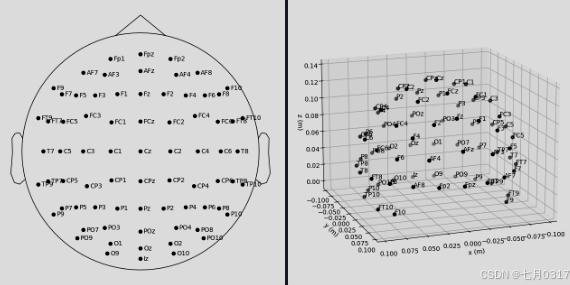
montage对象有一个在2D或3D中可视化传感器位置的绘图方法:
1 | easycap_montage.plot() # 2D |
加载后,可以使用set_montage方法将蒙太奇应用于数据,例如raw. set_montage(),epochs. set_montage()。这只适用于eeg通道名称对应于montage中的那些数据。
可以通过plot_sensors()方法可视化传感器位置。通过将montage名称直接传递给set_montage()方法,可以跳过手动蒙太奇加载步骤。
这里我们加载了一些非常规的sample数据,以展示手动用已有的文件设置montage,适用于eeg通道名称和内置montage不对应的情况。
1 | ssvep_folder = mne.datasets.ssvep.data_path() |
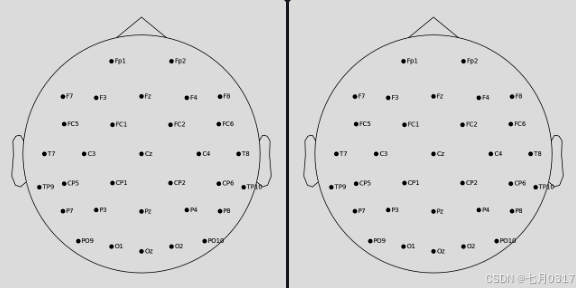
注意,通过plot_sensors()创建的图形可能比easycap_montage.plot()的结果包含更少的传感器。这是因为montage包含了为EEG系统定义的所有通道;但并不是所有的记录都必须使用所有可能的通道。因此,将montage应用于实际的EEG数据集时,有关数据中实际不存在的传感器的信息被删除。
像EEGLAB一样绘制2D传感器位置
如果你喜欢使用10-20个conventions来绘制头部圆圈,可以传递sphere=’ EEGLAB ‘,注意,因为我们在这里使用的数据不包含Fpz频道,所以它的假定位置是自动近似的。:
1 | fig = ssvep_raw.plot_sensors(show_names=True, sphere="eeglab") |
手动控制2D通道投影
通道在2D空间中的位置是通过将其实际的3D位置投影到球体上,然后将球体投影到平面上来获得的。默认情况下,使用原点为(0,0,0),半径为0.095米(9.5厘米)的球体。你可以通过在任何绘制2D通道的函数中传递单个值作为sphere参数来使用不同的球体半径:
1 | fig1 = easycap_montage.plot() # default radius of 0.095 |
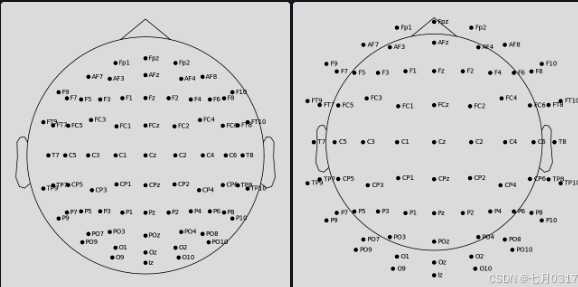
为了不仅改变半径,还改变球体原点,传递一个(x, y, z, radius)元组作为球体参数
1 | fig = easycap_montage.plot(sphere=(0.03, 0.02, 0.01, 0.075)) |
读取传感器数字文件
在示例数据中,传感器位置已经在Raw对象的info属性中可用。因此,我们可以使用plot_sensors()直接从Raw对象绘制传感器位置,它提供了与montage.plot()相似的功能。
此外,plot_sensors()支持按类型选择通道,默认情况下,raw.info[‘bads’]中列出的通道将被绘制为红色,并在现有的Matplotlib Axes对象中绘制,因此通道位置可以很容易地作为多面板图中的子图添加:
1 | sample_data_folder = mne.datasets.sample.data_path() |
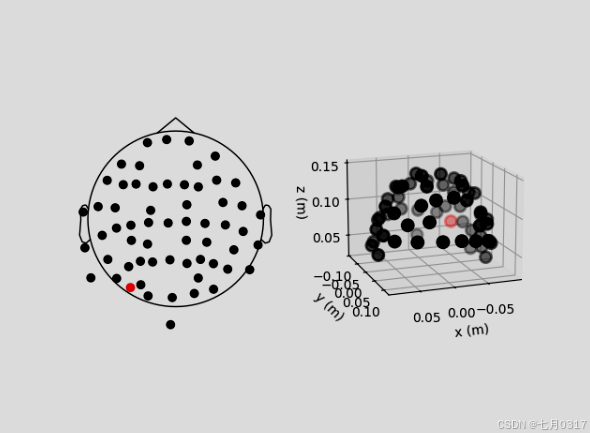
注意,当使用set_montage()设置montage时,测量信息在两个地方更新(chs和dig条目都被更新)除了电极位置外,dig还可能包含HPI、基准点或头部形状点信息。
传感器3D表面渲染的可视化
用一个新函数,如下所示:
1 | fig = mne.viz.plot_alignment( |

注意,plot_alignment()需要一个Info对象,并且还可以渲染头皮,头骨和大脑的MRI表面(通过将带有’head’, ‘outer_skull’或’brain’键名的字典传递给surfaces参数)。这使得该函数对于评估坐标坐标系转换很有用。
关于layout文件的使用
与montage类似,许多layout文件都包含在MNE-Python中,它们存储在mne/channels/data/layouts文件夹中:
1 | layout_dir = Path(mne.__file__).parent / "channels" / "data" / "layouts" |
BUILT-IN LAYOUTS
CTF-275.lout
CTF151.lay
CTF275.lay
EEG1005.lay
EGI256.lout
GeodesicHeadWeb-130.lout
GeodesicHeadWeb-280.lout
KIT-125.lout
KIT-157.lout
KIT-160.lay
KIT-AD.lout
KIT-AS-2008.lout
KIT-UMD-3.lout
Neuromag_122.lout
Vectorview-all.lout
Vectorview-grad.lout
Vectorview-grad_norm.lout
Vectorview-mag.lout
biosemi.lay
magnesWH3600.lout
要加载布局文件,可以使用mne.channels.read_layout函数。然后可以使用它的plot方法可视化布局:
与用于从Raw对象中选择通道的picks参数类似,Layout对象的plot()方法也有一个picks参数。然而,由于布局只包含有关传感器名称和位置信息,没有类型信息,所以只支持按索引选择通道,而不是按名称或类型。在下面的示例中,我们使用numpy.where()找到所需的索引;使用mne.pick_channels()或mne.pick_types()按名称或类型进行选择。
1 | biosemi_layout = mne.channels.read_layout("biosemi") |
1 | midline = np.where([name.endswith("z") for name in biosemi_layout.names])[0] |

如果你有一个包含传感器位置的Raw对象,你可以用mne.channels.make_eeg_layout()或mne.channels.find_layout()创建一个Layout对象。
1 | layout_from_raw = mne.channels.make_eeg_layout(sample_raw.info) |

注意,没有相应的make_meg_layout()函数,因为传感器位置在MEG系统中是固定的,不像在EEG中,传感器帽会变形以紧贴特定的头部。因此,对于给定的系统,MEG布局是permanent的,所以可以简单地使用mne.channels.read_layout()加载它们,或者使用带有ch_type参数的mne.channels.find_layout()如前面为EEG演示的那样)。